Validación taxonómica - Taxonomic Name Resolution Service (TNRS) para plantas
Objetivo
Realizar una validación taxonómica automática de los nombres científicos de plantas con TNRS.
Introducción
Convenciones
- Los elementos del estándar Darwin Core aparecen en color verde y cursiva. Por ejemplo: measurementUnit.
- Los archivos a utilizar en los ejercicios se muestran entre comillas angulares, negrita, y tienen una tipografía diferente. Por ejemplo: «archivo_Ejemplo.xls».
- Las secciones, ventanas y componentes de las herramientas utilizadas se muestran entre comillas inglesas y en negrita. Por ejemplo: “Create Project”.
- Las opciones de las herramientas que se asocian a instrucciones (dar clic, seleccionar, etc.) aparecen en color amarillo y cursiva. Por ejemplo: New project.
- Las secuencias de instrucciones y pasos se muestran en color amarillo, cursiva y negrita. Por ejemplo: Paso 1 > Paso 2.
- Las líneas que se escriben directamente en las herramientas, para programar o realizar algún proceso en específico, aparecen en formato de código, con una tipografía distinta de color negro. Por ejemplo: value.replace(“ sp.”,””).
Sobre la herramienta
La herramienta TNRS, por su sigla en inglés (Taxonomic Name Resolution Service), es una aplicación originalmente desarrollada por iPlant que permite estandarizar los nombres científicos botánicos a partir de fuentes taxonómicas como Tropicos, USDA y TPL. La validación taxonómica con TNRS sirve para:
- Identificar errores ortográficos o de tipeo.
- Separar el nombre científico en diferentes elementos Darwin Core (genus, specificEpithet, scientificNameAuthorship).
- Evaluar la coincidencia del nombre científico, familia, género, epiteto específico e infraespecífico y autoría, al comparar los datos con las fuentes taxonómicas de referencia.
- Listar otras posibles coincidencias de un nombre científico con su respectivo enlace a la fuente.
- Identificar sinonimias y nombres actualmente aceptados.
Enlace
Taxonomic Name Resolution Service (TNRS): https://tnrs.biendata.org/
Requerimientos
- Para realizar este ejercicio, es necesario contar con un procesador de archivos de texto como Excel.
Archivo de trabajo
Descargue el archivo «Datos_TNRS.xlsx» para realizar el laboratorio.
Paso 1 - Ingreso
Ingrese a la aplicación TNRS y familiarícese con las opciones. La herramienta está divida en dos partes: la primera (Fig. 1A) parmite colocar la lista de nombres científicos; la segunda (Fig. 1B) permite seleccionar las configuraciones.
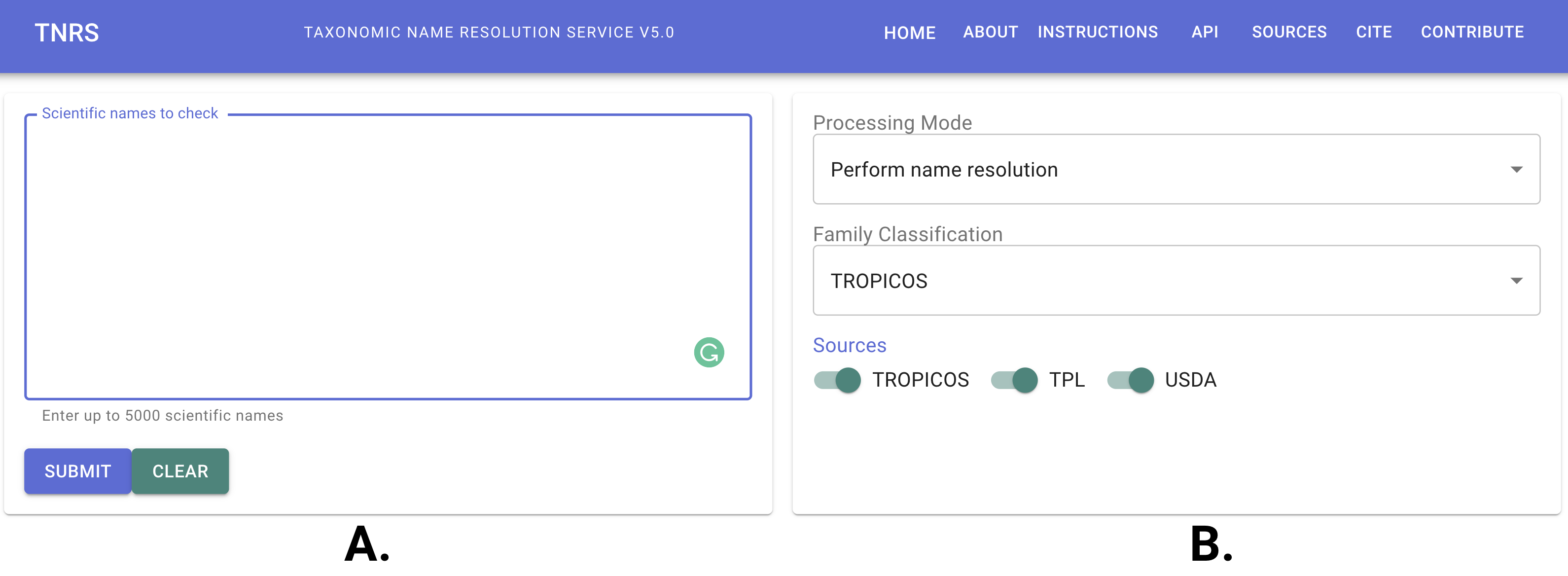
Figura 1. Descripción de las opciones de la herramienta: A. Lista de nombres científicos a validar, B. Configuraciones de la herramienta.
Paso 2 - Carga de los datos
Ingrese al archivo «Datos_TNRS.xlsx», seleccione la columna scientificName y copie la totalidad de esta sin incluir el encabezado.
Diríjase a la herramienta de TNRS y pegue los datos previamente copiados en la sección “Scientific names to check” (Fig. 1A). Asegúrese de que haya un solo nombre por línea y haga clic en SUBMIT (Fig. 2).
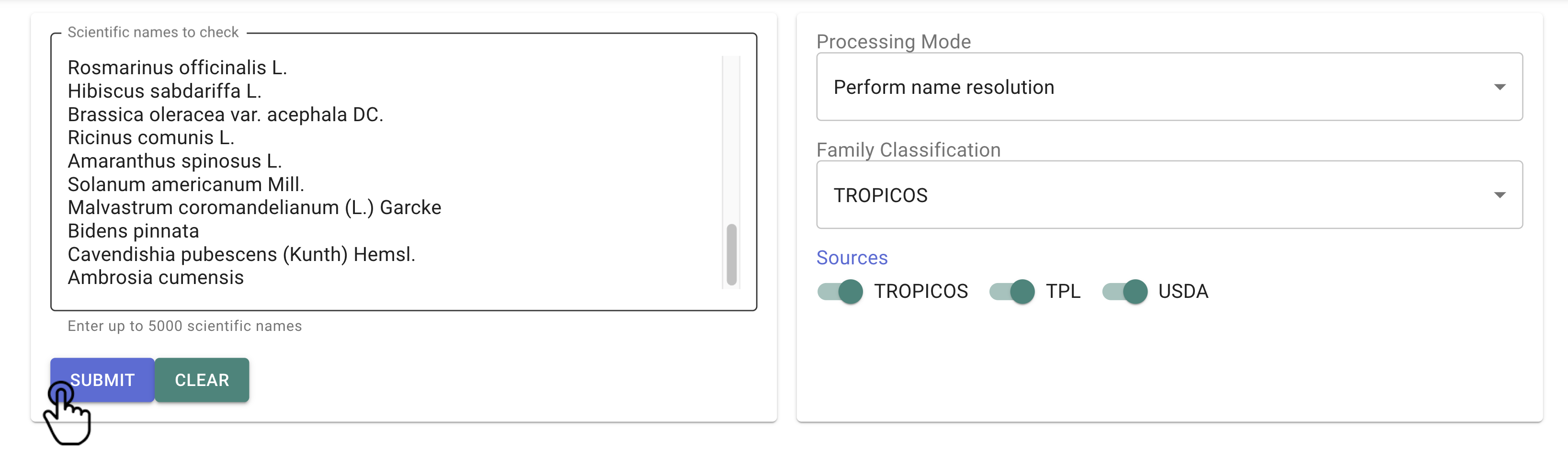
Figura 2. Carga de datos en la herramienta.
Paso 3- Selección de configuraciones
3.1. Modo de procesamiento
En la configuración “Processing Mode” (Modo de procesamiento), tiene dos opciones:
- Perform name resolution (Ejecutar resolución de nombres): separa el nombre científico en sus componentes y los valida con base en una o varias fuentes (resolución).
- Parse names only (Separar nombres únicamente): separa el nombre científico en sus componentes sin evaluar la coincidencia del nombre con las fuentes.
Para efectos de este ejercicio, seleccione la opción Perform name resolution.
3.2. Clasificación
La configuración de “Family Classification” permite seleccionar las familias según la clasificación APG IV. En este momento, solo está disponible la opción TROPICOS. Sin embargo, otras opciones pueden ser agregadas a futuro.
3.3. Fuente
En la configuración de “Source”, puede elegir las fuentes base para contrastar los datos. Es posible elegir una o varias fuentes:
Mantenga todas las fuentes seleccionadas. Por último, haga clic en el botón submit para iniciar la validación y espere a que se generen los resultados.
Paso 4 - Revisión preliminar de resultados
Esta revisión se realiza directamente en la herramienta, donde verá una tabla con los resultados del proceso si la ejecución fue exitosa y algunas opciones de configuración adicionales (Fig 3).
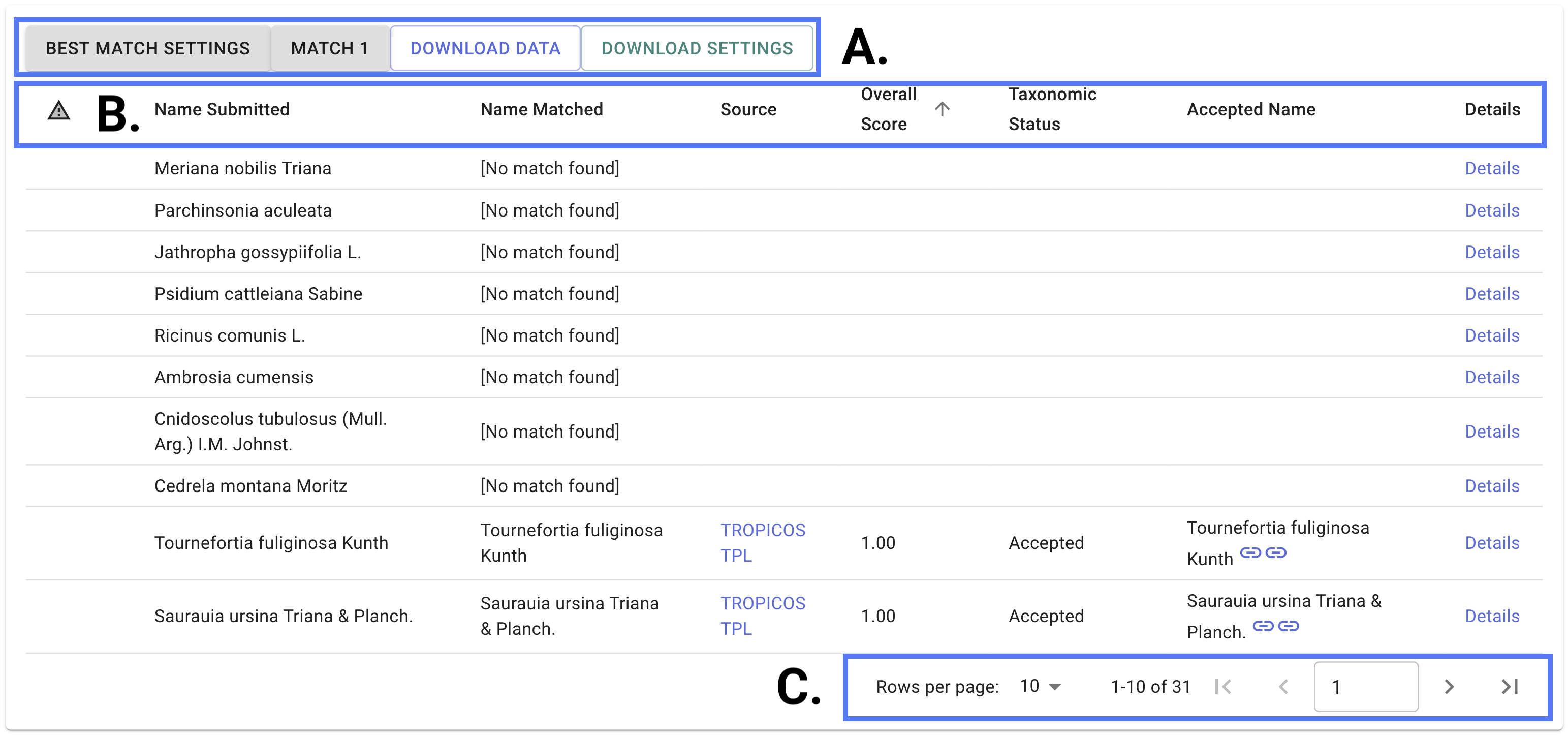
Figura 3. Configuraciones presentes en la revisión preliminar: A. Opciones adicionales de configuración, B. Columnas para la revisión de los resultados de validación, C. Paginación de los resultados.
4.1. Configuraciones adicionales
Después de realizar la validación, se activan algunas opciones adicionales para filtrar los resultados, cambiar el puntaje de coincidencia y descargar la validación.
- BEST MATCH SETTINGS: filtra los resultados según el puntaje de coincidencia (Overall Score) o según la taxonomía superior (Higher Taxonomy).
- MATCH: permite cambiar el puntaje de coincidencia (Match) de la validación. Entre más alto sea (cercano o igual a 1), la coincidencia tendrá que ser más exacta respecto a la fuente. Entre más cercano a 0, la coincidencia será menos precisa y le mostrará más resultados posibles de contraste (Fig 4).
Para este ejercicio, lo ideal es dejar el valor por defecto (SET DEFAULT), el cual corresponde a 0.53.
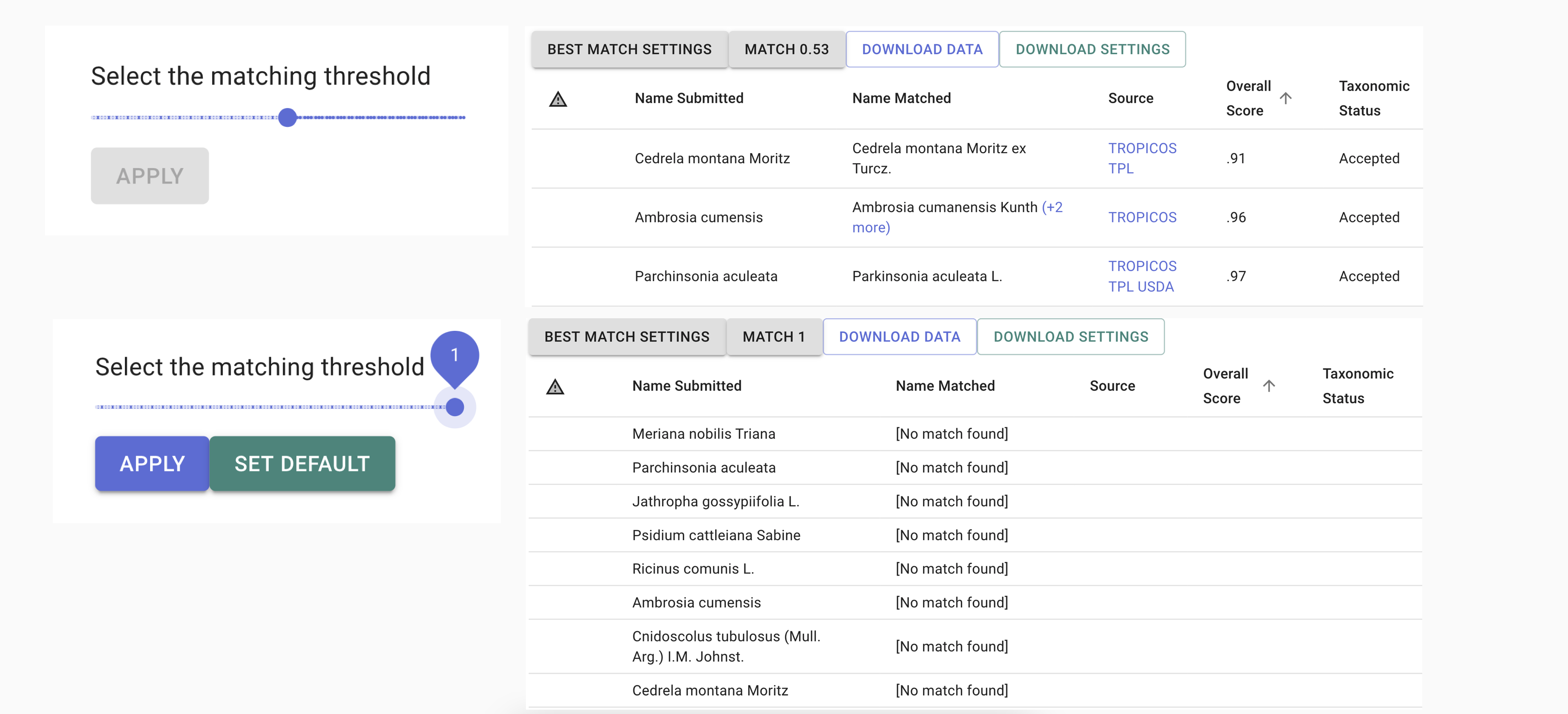
Figura 4. Cambio en el valor de MATCH.
-
DOWNLOAD DATA: descarga de los resultados. Al hacer clic en esta opción, una ventana emergenten mostrará las opciones de descarga (se abordará detalladamente en el paso 5).
-
DOWNLOAD SETTINGS: se descarga un archivo de texto plano con todas las configuraciones de la validación.
4.2. Tabla de resultados
Los resultados se muestran en 8 columnas, las cuales se enlistan a continuación:
Puede organizar la información de forma ascendente o descendente, haciendo clic en el nombre de cada columna (Fig. 3B).
-
“Warnings”: señala las inconsistencias del nombre validado a partir de un ícono de alerta (Fig 5). Haga clic en el símbolo para obtener una explicación adicional del problema. Las alertas más comunes son:
- Ambiguos match: hay más de un nombre sugerido con el mismo puntaje de coincidencia y estado taxonómico.
- Partial match: el nombre sugerido pertenece a una catageoría taxonómica superior a la del nombre original.
- Better higher taxonomic match available: existe otro nombre que tiene mejor coincidencia con la taxonomía superior.
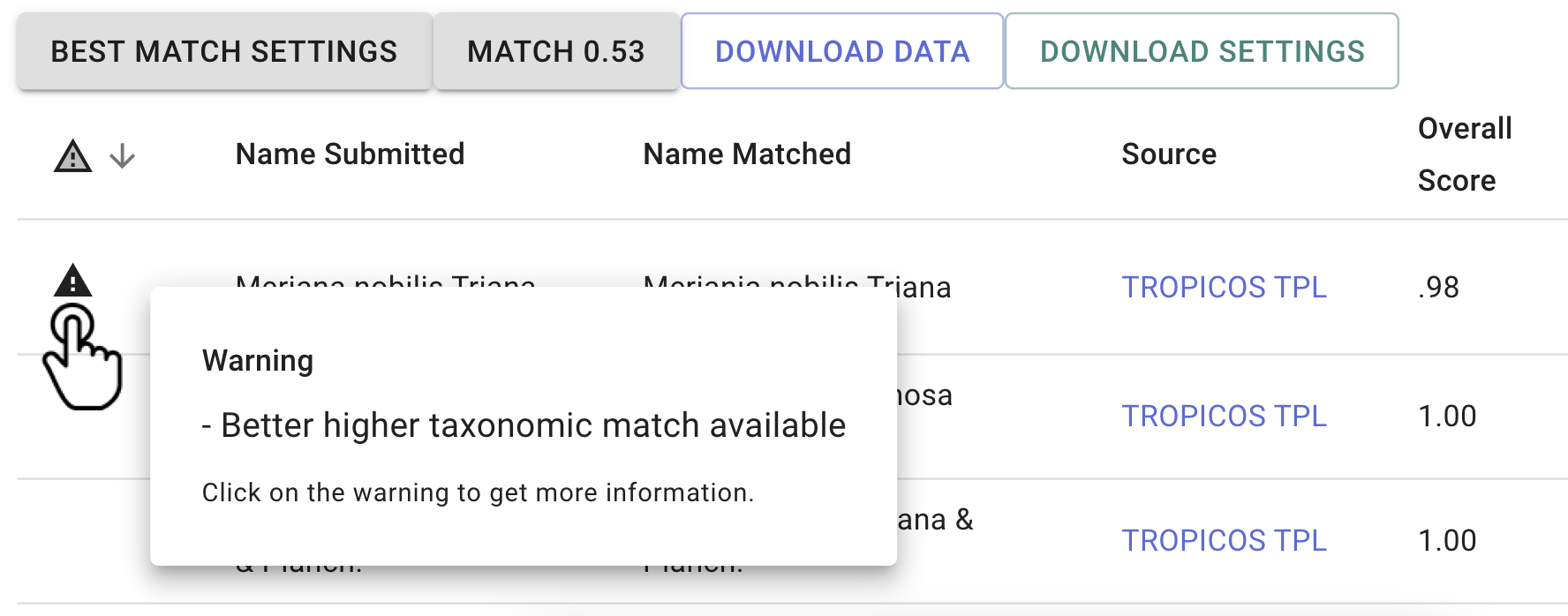
Figura 5. Ejemplos de problemas.
- “Name submitted”: el nombre científico original ingresado en la herramienta.
- “Name Matched”: Nombre científico con el mejor puntaje de coincidencia. En esta columna, puede encontrar un mensaje +n more. La expresión +n es el número de posibles coincidencias encontradas para ese nombre. Dé clic al botón +n more para ver la pestaña adicional, donde puede seleccionar qué taxonomía prefiere.
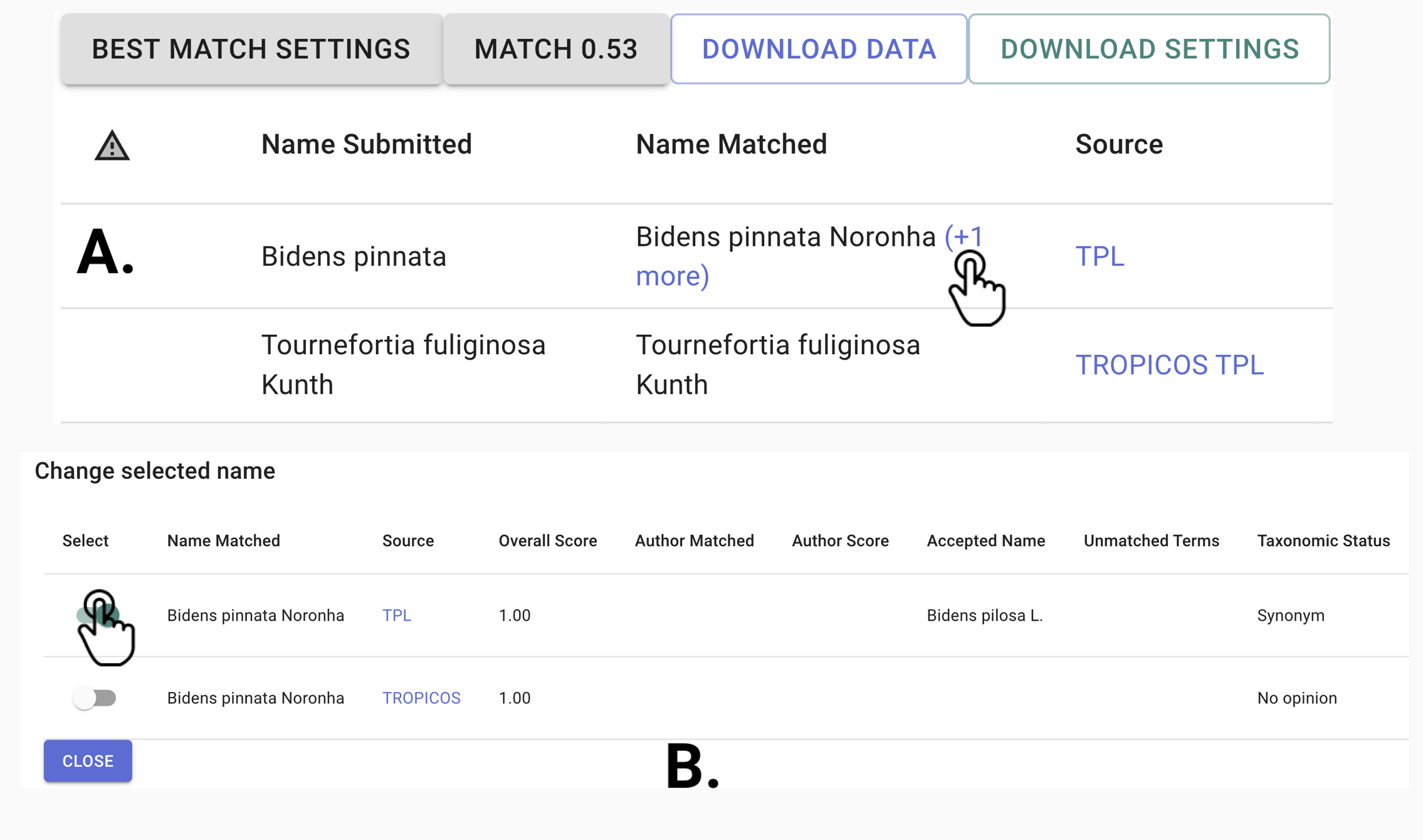
Figura 6. Cambio en el tipo de taxonomía.
- “Source”: la fuente donde se encontró el nombre con el mejor puntaje.
- “Overall Score”: un puntaje que indica la similitud entre el nombre original y el encontrado, donde 1 es el valor mas alto de coincidencia.
- “Taxonomic Status”: el estado taxonómico del nombre con el mejor puntaje. Por ejemplo, aceptado, inválido, sinónimo, etc.
- “Accepted Name”: el nombre canónico aceptado para el taxón.
- “Details”: muestra mayor detalle de la validación a partir de columnas con información adicional.
Finalmente, se puede modificar el número de filas visualizadas por página y navegar entre las diferentes páginas (Fig. 3C).
Paso 5 - Descarga de resultados
Haga clic en el botón DOWNLOAD DATA. Posteriormente, encontrará las siguientes secciones en la ventana emergente:
-
“File Name”: nombre del archivo de descarga. Por defecto, aparece tnrs_result.
-
“Download format”: formato de descarga del archivo. Esta sección brinda dos opciones:
- CSV: del inglés “Comma separted value”, es un archivo de texto plano separado por comas (,).
- TSV: del inglés “Tab separted value”, es un achivo de texto plano separado por tabuladores.
-
Results to Download: permite seleccionar el tipo de resultado a descargar. Esta sección brinda dos opciones:
- Best Matches Only: en caso de que un nombre científico tenga varias coincidencias, se descargará la alternativa con el puntaje de coincidencia más alto.
- All Matches: en caso de que un nombre científico tenga varias coincidencias, se descargan todas las alternativas para dicho nombre (+n more).
Configure las descargas como se muestra en la Figura 7 y haga clic en DOWNLOAD.
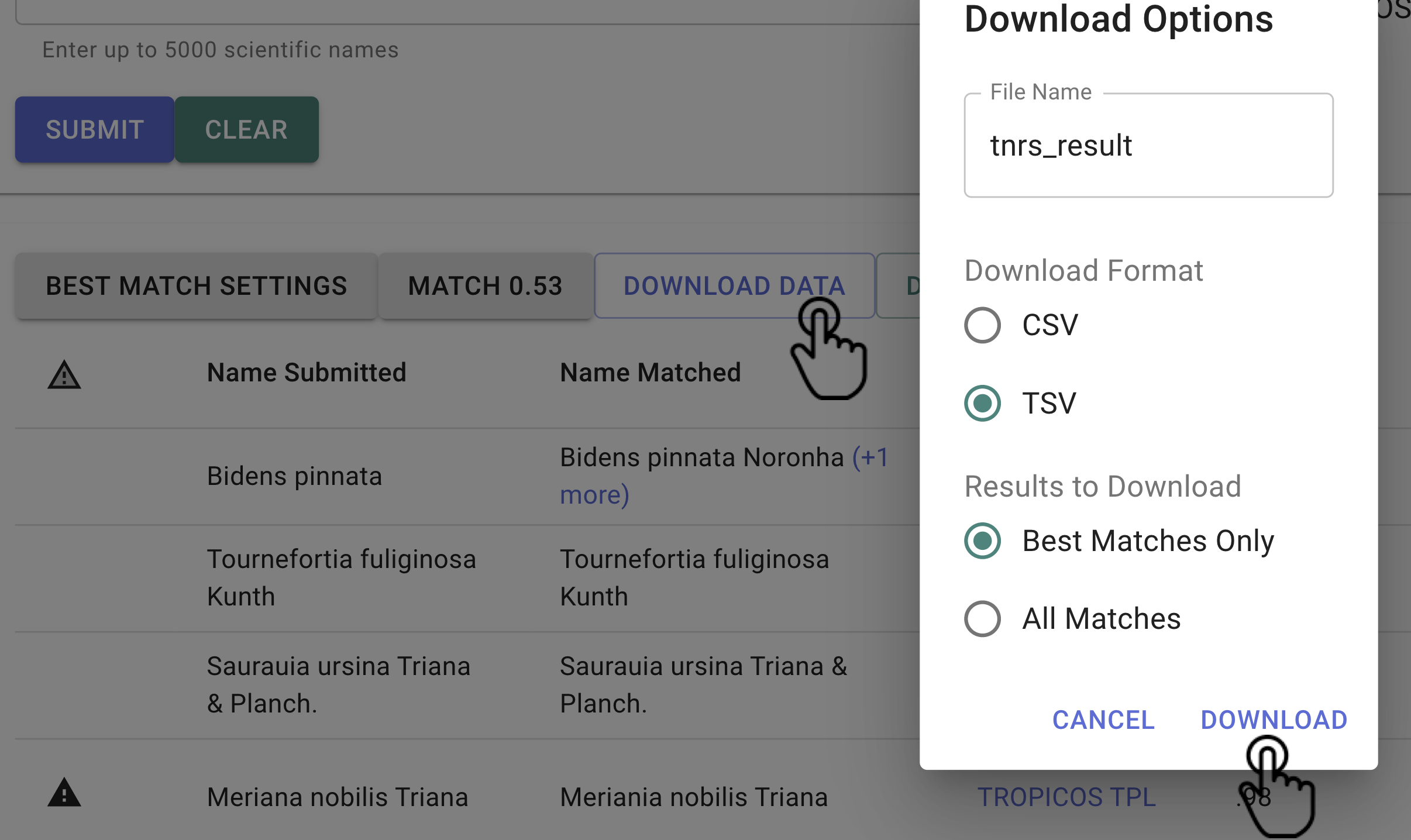
Figura 7. Descarga de los datos.
Paso 6 - Visualización de resultados
Los resultados se descargarán como un archivo de texto plano. Por consiguiente, puede abrirlos en un editor de texto como Excel u OpenRefine. Si necesita una guía de cómo abrir este tipo de archivo en Excel, lo invitamos a consultar la Guía de importación de resultados en Excel.
Paso 7 - Revisión de resultados y modificación de datos
El archivo de la validación tiene un total de 47 columnas. Entre estas, se encuentran las 7 columnas vistas en la previsualización de resultados y varias columnas adicionales que indican la taxonomía obtenida.
A continuación, se describen algunas de las columnas adicionales que encontrará en la descarga para validar los resultados con mayor detalle:
- “Author_matched”: autoría del nombre científico con el mayor puntaje de coincidencia.
- “Genus_submitted”: género ingresado.
- “Genus_matched”: género con el mejor puntaje de coincidencia.
- “Specific_epithet_submitted”: epíteto específico ingresado.
- “Specific_epithet_matched”: género con el mayor puntaje de coincidencia.
- “Accepted_name”: nombre canónico aceptado para el taxón.
- “Accepted_name_author”: autor del nombre canónico aceptado para el taxón.
- “Accepted_family”: familia aceptada para el nombre con el mayor puntaje.
- “Accepted_name_rank”: categoría taxonómica del nombre aceptado del taxón.
- “Source”: fuente donde se encontró el nombre con el mayor puntaje.
- “Warnings”: si no hay coincidencias, aparecerá una alerta.
- “Accepted_name_lsid”: identificador del nombre aceptado para el taxón, el cual se obtiene solo cuando la fuente es GCC.
Luego de familiarizarse con los resultados de la tabla, realice los siguiente ajustes en el archivo «Datos_TNRS.xlsx»:
-
Identifique el nombre científico validado como una sinonimia y realice los respectivos ajustes. Tenga en cuenta revisar también los elementos genus y specificEpithet si realizó cambios en scientificName.
-
En los géneros hay 3 errores de tipeo, identifiquelos y ajústelos en el elemento genus.
-
En los epítetos específicos hay 3 errores de tipeo, identifiquelos y ajústelos en el elemento specificEpithet.
-
Ajuste los nombres científicos (scientificName), de acuerdo a los anteriores cambios.
-
Complete las autorias de los nombres cientítificos en el elemento scienficNameAuthorship.
-
Ajuste las categorías taxonómicas al nombre científico validado en el elemento taxonRank, según corresponda.
¿Identificó otros ajustes a realizar en el conjunto de datos a partir de la validación?
Paso 8 - Verificación del resultado
Descargue y compare el siguiente archivo, validado según las definiciones del estándar Darwin Core, con el archivo que trabajó en el laboratorio e identifique aciertos y oportunidades de mejora.
¿Qué diferencias encontró con sus resultados?
¡Felicitaciones! Terminó la revisión de los nombres científicos con la herramienta TNRS.
Atribución y uso de los laboratorios

La licencia CC-BY permite usar, redistribuir y construir sobre estos contenidos libremente.
¡La difusión de estos laboratorios contribuirá a la publicación de más y mejores conjuntos de datos sobre biodiversidad!
Citación sugerida
Plata C., Ortíz R., Marentes E., Lozano J. (2021). Laboratorio de datos, Ciclo de formación. Consultado a través del SiB Colombia. Disponible en https://biodiversidad.co/formacion/laboratorios.
Referencias
Boyle, B., Hopkins, N., Lu, Z., Garay, J. A. R., Mozzherin, D., Rees, T., Matasci, N., Narro, M. L., Piel, W. H., McKay, S. J., Lowry, S., Freeland, C., Peet, R. K., & Enquist, B. J. (2013). The taxonomic name resolution service: an online tool for automated standardization of plant names. BMC Bioinformatics, 14(1), 16. https://doi.org/10.1186/1471-2105-14-16.