¡Precaución!
No utilice Internet Explorer para este laboratorio, ya que OpenRefine no funciona bien con este navegador web.
Objetivo
Profundizar en el uso de OpenRefine para validar de datos sobre biodiversidad de manera automática, utilizando las APIs de varias herramientas en línea.
Introducción
Próximamente, video introductorio.
Sobre la Herramienta
OpenRefine es un software creado con el objetivo de pulir datos crudos hasta convertirlos en diamantes, los cuales son activos valiosos en la era del BigData.
Este programa permite visualizar y manipular datos tabulares, facilitando el mejoramiento de la calidad general de un conjunto de datos. Tiene la apariencia de un software tradicional de hoja de cálculo (similar a Excel), pero funciona como una base de datos. Esto significa que OpenRefine no es adecuado para adicionar nuevas filas de datos, pero es extremadamente poderoso cuando se trata de explorar, limpiar y vincular datos.
OpenRefine es un software de código abierto bajo una licencia BSD y se instala localmente, por lo que funciona como una aplicación web personal y de acceso privado a la que se accede desde un navegador web.
Esta herramienta sirve para todo tipo de datos. Sin embargo, en este laboratorio se explica su funcionamiento en el contexto de datos sobre biodiversidad, estandarizados en Darwin Core. Puede consultar más información de la herramienta en el manual de usuario (en inglés).
Para saber más acerca de GREL, el lenguaje de programación de OpenRefine que se utiliza en las rutinas, consulte el manual de usuario (en inglés).
Convenciones
Esta guía adapta y profundiza en los procesos correspondientes a la sección 5 de la Guía para la limpieza de datos sobre biodiversidad con OpenRefine.
Requerimientos
Las rutinas comparan la información documentada en el conjunto de datos con diferentes fuentes de referencia y crean columnas de validación a partir de dicha comparación. En estas columnas se puede evidenciar la correspondencia entre el archivo original y la fuente de referencia a través de operadores lógicos, unos (1) y ceros (0), que funcionan como indicadores de validación.
Los indicadores de validación se interpretan así:
0: el valor documentado en el conjunto de datos NO coincide con la fuente de referencia, así que el valor debe ser revisado y ajustado en caso de ser necesario.
1: el valor documentado en el conjunto de datos coincide con la fuente de referencia, así que no es necesario tomar acciones adicionales.
Observe el ejemplo de la Figura 1. En la primera fila, el valor original de la columna family no coincide con la columna “familySuggested”¨, ya que tiene un error de tipeo. Por lo tanto, el indicador de validación (columna “familyValidation”) es cero (0). Note que el indicador de validación (“familyValidation”) de las filas donde sí hay coincidencia es uno (1).
Las rutinas utilizan API’s (Interfaces de Programación de Aplicaciones) de repositorios globales taxonómicos como fuentes de validación. Las API’s pueden ser de carácter geográfico o archivos de texto plano obtenidos a partir de herramientas externas de validación.
Hay seis rutinas disponibles (Tabla 1), en esta guía se profundizará en 4 de ellas.
Tabla 1. Lista de rutinas para la validación de datos primarios sobre biodiversidad.
| Nombre | Uso | Requerimientos |
|---|---|---|
| Validación taxonómica con el API de GBIF | Validación taxonómica que usa como referencia el árbol taxonómico de GBIF. Permite validar registros de varios grupos biológicos a la vez, así como obtener la taxonomía superior de cada taxa. | Requiere como mínimo la documentación de los elementos DwC scientificName y kingdom y acceso a Internet para hacer la solicitud al API de GBIF. |
| Validación taxonómica con Species Matching de GBIF | Validación taxonómica que usa como referencia el árbol taxonómico de GBIF. A diferencia de la rutina anterior, realiza la validación con base en el archivo de resultados «normalized», obtenido de Species Matching. Por lo tanto, permite aprovechar las funcionalidades de validación y limpieza de esta herramienta. La rutina facilita el cruce de los resultados obtenidos con Species Matching respecto al conjunto de datos original. | Requiere como mínimo que el elemento DwC scientificName esté documentado y que el archivo «normalized» sea previamente cargado en OpenRefine para ejecutar la rutina. |
| Validación taxonómica con el API de WoRMS | Validación taxonómica específica para organismos marinos, que usa como referencia el árbol taxonómico de LifeWatch (LW-SIBb) por medio de la API de WoRMS (World Register of Marine Species). Permite obtener la taxonomía superior de cada taxa, así como elementos taxonómicos obligatorios para la publicación de datos a través de OBIS. | Requiere como mínimo la documentación del elemento DwC scientificName y acceso a Internet para hacer la solicitud al API de GBIF. |
| Validación de elevaciones con el API de GeoNames | Validación y obtención de la elevación a partir de las coordenadas con el servicio geográfico de GeoNames. | Requiere como mínimo la documentación de los elementos DwC decimalLatitude y decimalLongitude y acceso a Internet para hacer la solicitud al API de GeoNames. |
| Validación de nombres geográficos con DIVIPOLA | Validación de los nombres oficiales de departamentos, municipios y centros poblados | Requiere como mínimo que el elemento DwC stateProvince (departamento) esté documentado. Adicionalmente, valida los elementos county (municipio) y municipality (centro poblado) si están documentados. |
| Transformación de fechas con el API de Canadensys | Transformación de fechas en múltiples formatos al estándar ISO 8601. | Requiere la documentación del elemento DwC eventDate y acceso a Internet para hacer la solicitud al API de Canadensys. |
Las rutinas cuya fuente de referencia es un API consultan a un servicio externo y obtienen una respuesta en formato JSON. Cada rutina interpreta esta respuesta y la hace legible en forma de columnas dentro del conjunto de datos. Posteriormente, el resultado de la consulta al API es comparado con el valor documentado en el conjunto de datos y se generan nuevas columnas con los indicadores de la validación (unos y ceros).
Las rutinas que usan como fuente archivos de texto plano consultan con base en un archivo subido previamente a OpenRefine, el cual es comparado con el valor documentado en el conjunto de datos. Como resultado de la comparación se generan nuevas columnas con los indicadores de la validación.
Todas las rutinas se ejecutan de manera similar, los detalles específicos para cada una se explican más adelante. Esta guía se enfoca en 4 rutinas: dos taxonómicas y dos geográficas, por lo que se divide en dos partes.
La primera parte de la guía aborda la validación taxonómica con el API de GBIF y WoRMS, lo cual permite consultar estos árboles taxonómicos de manera directa, sin utilizar las aplicaciones en línea Species Matching o WoRMS TaxonMatch. Esta ruta directa también le permite hacer consultas para una mayor cantidad datos y elementos, sin los limitantes de las aplicaciones en línea.
Dependiendo de su interés particular, realice esta guía con alguna de las suguientes opciones:
Datos de grupos biológicos principalmente continentales
Datos de grupos biológicos marino-costeros
Cree un proyecto en OpenRefine con el conjunto de datos que desea validar. Si tiene dudas sobre cómo hacerlo, revise el paso 2 de la Guía general de OpenRefine.
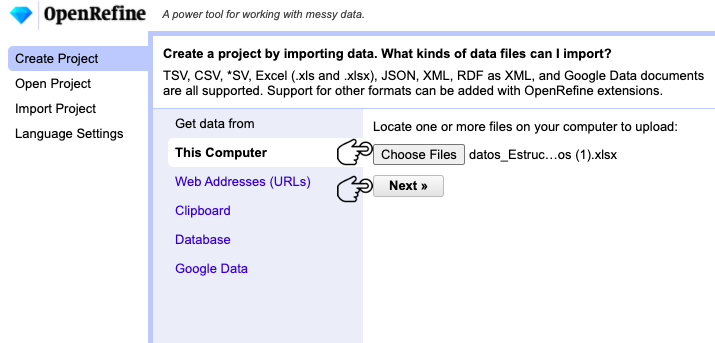
Figura 1 . Creación de un nuevo proyecto en OpenRefine
Asegúrese de que el conjunto de datos cumpla con los elementos y requerimientos mínimos de cada rutina:
Validación taxonómica con el API de GBIF - Datos continentales
Requiere como mínimo la documentación de los elementos DwC scientificName y kingdom y acceso a Internet para hacer la solicitud al API de GBIF.
Haga un Text facet en los elementos kingdom y class (Fig. 2).
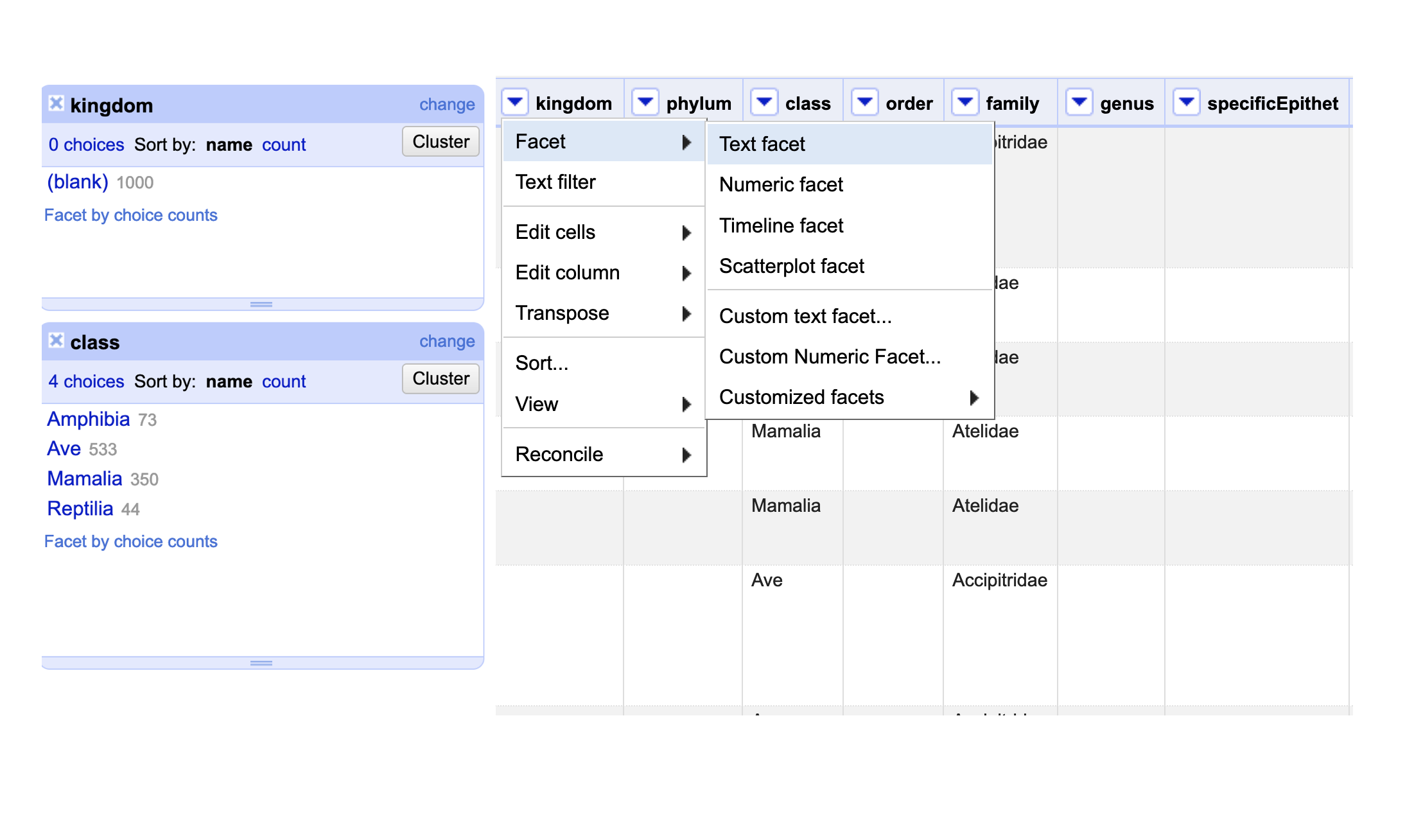
Figura 2. Text facet en los elementos kingdom y class.
Observe que el conjunto de datos no tiene documentado el elemento kingdom para ningún registro. Sin embargo, a partir del filtro sobre el elemento class, se puede inferir que todos los datos corresponden a animales. Por consiguiente, complete el reino para todos los registros (Fig. 3).
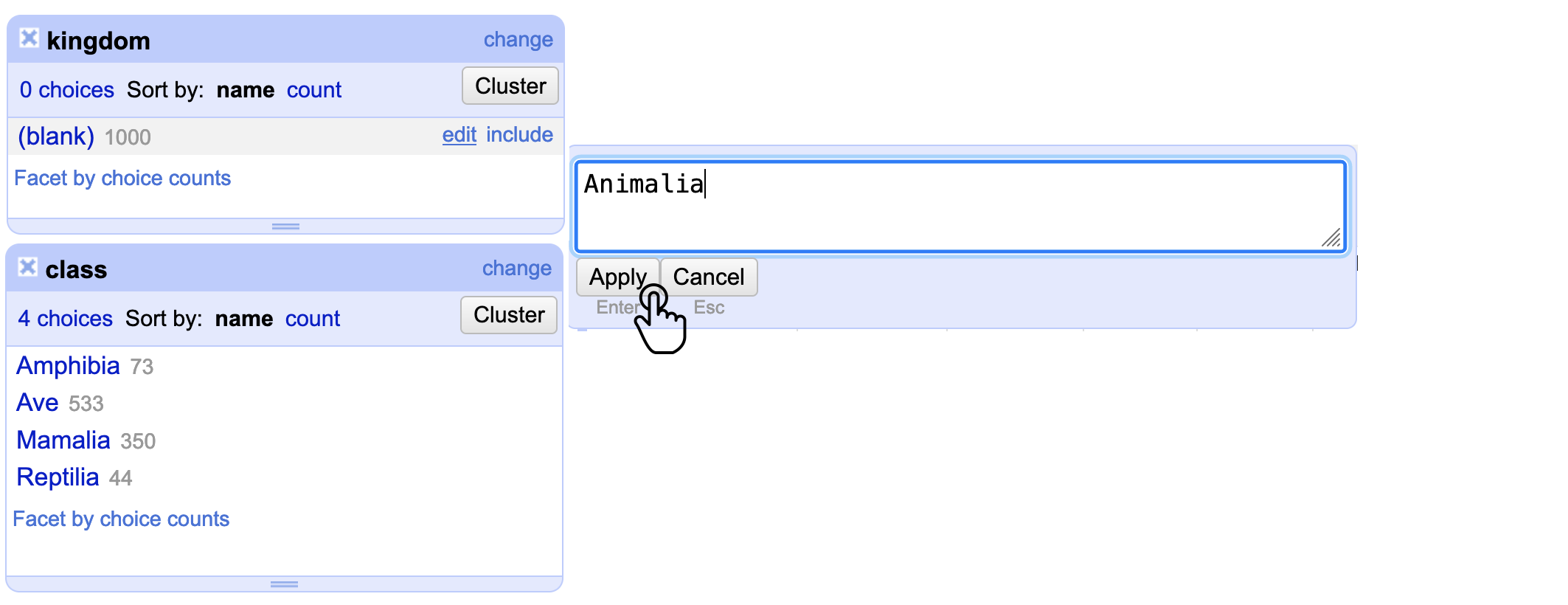
Figura 3. Ajuste previo a correr la rutina del elemento kingdom.
Haga una limpieza previa del elemento scientificName para obtener mejores resultados:
Validación taxonómica con el API de WoRMS - Datos marino-costeros
Requiere como mínimo la documentación del elemento DwC scientificName y acceso a Internet para hacer la solicitud al API de GBIF.
Haga una limpieza previa del elemento scientificName para obtener mejores resultados. Puede ver las recomendaciones enteriores en la sección de “Datos continentales”.
Seleccione la rutina de interés según la validación que desee realizar:
Rutina de Validación taxonómica con el API de GBIF - Datos continentales.
Rutina de Validación taxonómica con el API de WoRMS - Datos marino-costeros.
Haga clic en el enlace a la rutina y será redirigido a GitHub, donde encontrará un archivo de texto plano con la rutina. Luego, copie el texto de la rutina de validación (Fig. 4). Asegúrese de seleccionar solo la rutina -sin las instrucciones- y copiar todos los corchetes iniciales ({) y finales (}), ya que estos son fundamentales para que la rutina se ejecute correctamente.
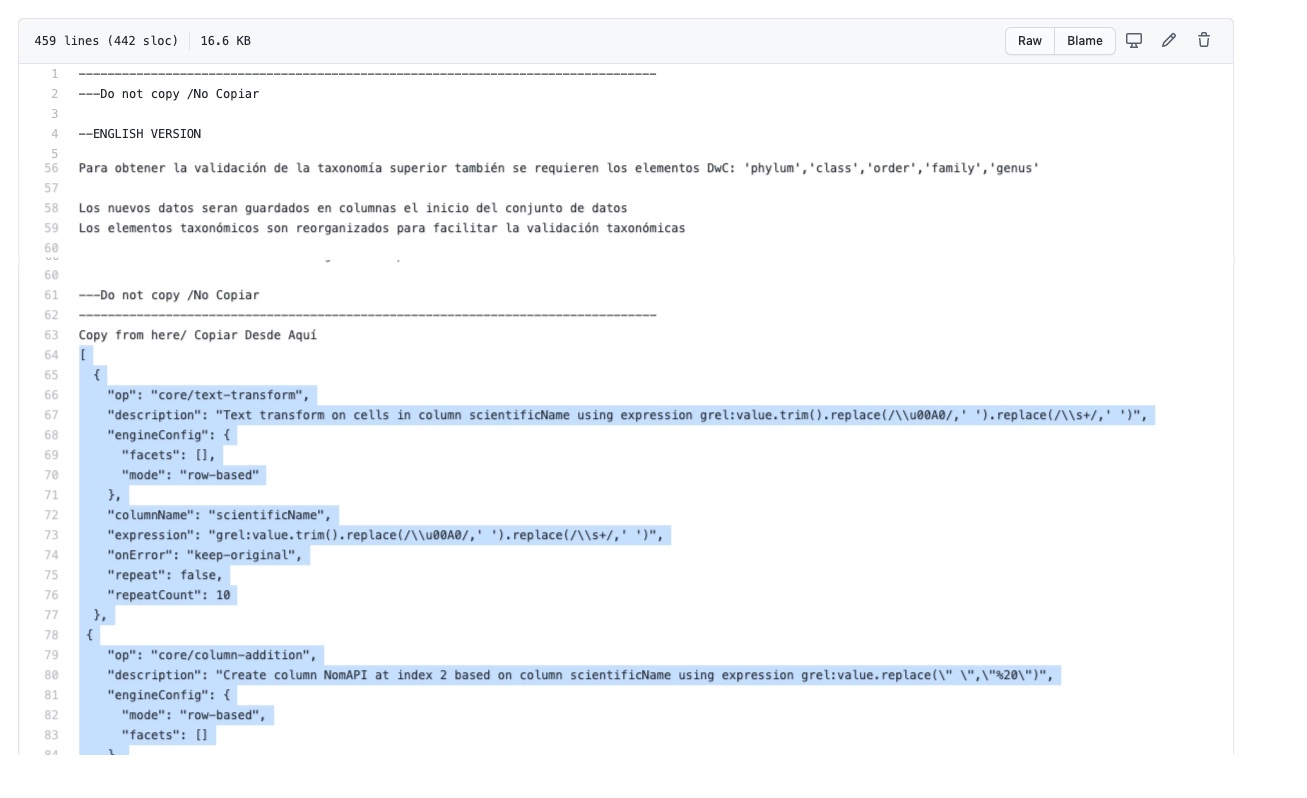
Figura 4. Selección de la rutina a ejecutar.
En el conjunto de datos a validar en OpenRefine, diríjase al menú superior izquierdo, seleccione la pestaña Deshacer/Rehacer y haga clic en el botón Aplicar…. A continuación, se abrirá una ventana de texto vacía. Pegue la rutina a ejecutar en el cuadro de texto y dé clic en Ejecutar Operaciones (Fig. 5).
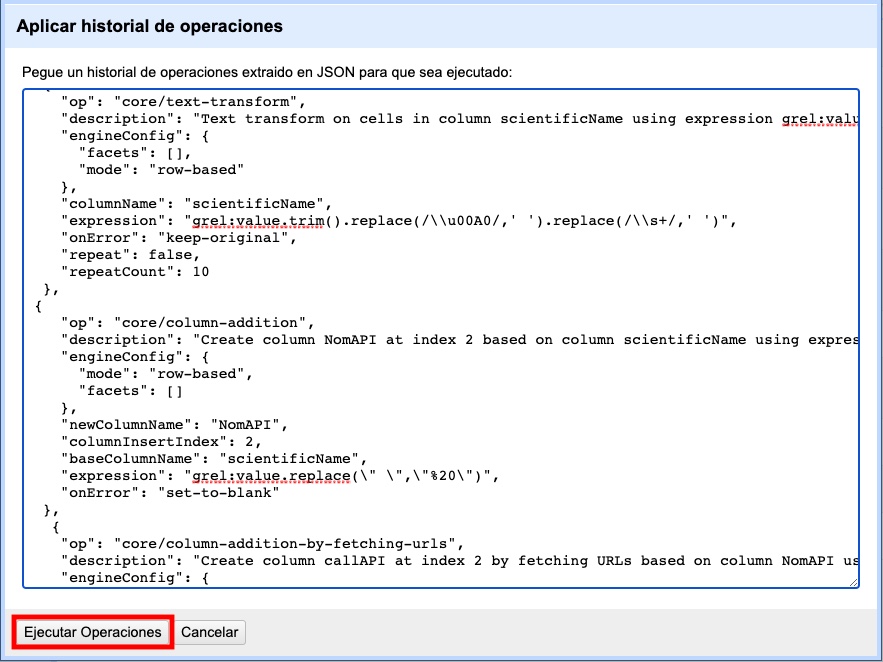
Figura 5. Ejecución de la rutina en OpenRefine.
El avance de la ejecución de la rutina se observa en la parte superior de la pantalla (Fig. 6).

Figura 6. Avance de la ejecución de la rutina en OpenRefine.
Espere a que finalice la ejecución de la rutina. Las rutinas que requieren hacer llamados a servicios externos dependen de la conexión a Internet. Por consiguiente, estas consultas toman un tiempo en ejecutarse, el cual varía según el número de filas del conjunto de datos, de la velocidad de la conexión y de la memoria RAM de su equipo.
Al terminar la ejecución de la rutina, obtendrá nuevas columnas en el conjunto de datos. Puede identificarlas por su terminación:
“Suggested”: son valores sugeridos por la validación con las fuentes de referencia. Dependiendo de la rutina seleccionada, pueden ser sugerencias taxonómicas, geográficas o temporales.
“Validation”: corresponde a los indicadores de validación (unos y ceros) que permiten rastrear diferencias entre el valor original y el valor sugerido para realizar posteriormente una limpieza de los datos.
En la Figura 7 se muestra un ejemplo de cómo se ven los identificadores de la validación y las nuevas columnas con las sugerencias después de ejecutar la rutina. El ejemplo muestra una validación taxonómica, pero las columnas de resultado pueden variar según el objetivo de cada rutina.
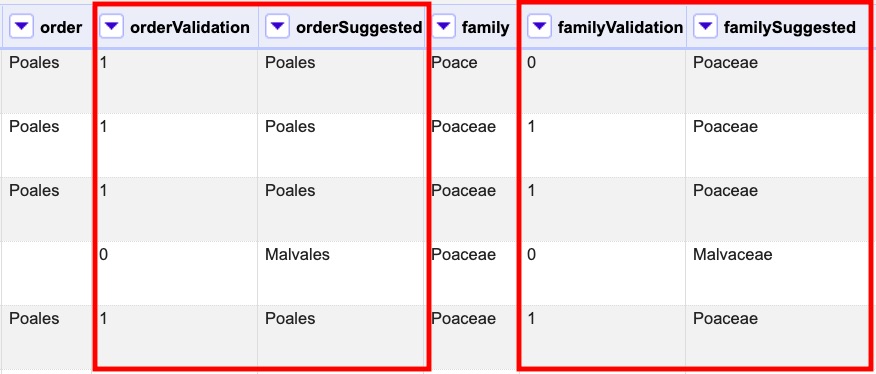
Figura 7. Identificadores de validación y columnas resultantes de la rutina.
4.1 Datos de grupos biológicos principalmente continentales
Funcionamiento:
Esta rutina valida la información taxonómica de un conjunto de datos, usando como referencia el árbol taxonómico de GBIF. Esto se logra a través de un llamado al API de GBIF con base en los elementos del estándar DwC scientificName y kingdom que se documentan en el conjunto de datos. Como resultado, el llamado retorna la taxonomía superior, nombres aceptados, estatus taxonómico y autoría del nombre científico de acuerdo al árbol taxonómico de GBIF. La rutina toma los valores obtenidos del árbol y los compara con los elementos documentados en el archivo base, generando los indicadores de validación que se explican a continuación.
Resultados:
En las primeras columnas del proyecto, encontrará los datos taxonómicos reorganizados junto con las nuevas columnas, resultado de la rutina. Primero encontrará las columnas asociadas al cruce con el árbol taxonómico y luego, de manera intercalada, las columnas con el valor taxonómico original, un valor sugerido de acuerdo al árbol taxonómico de GBIF y el indicador de validación que establece si los valores coinciden (1) o difieren (0).
A continuación, se listan las columnas que encontrará después de ejecutar la rutina:
“taxonMatchType”: indica el resultado del cruce de los datos originales con el árbol taxonómico de GBIF a partir de los elementos scientificName y kingdom. Los valores que encontrará en esta columna son:
EXACT: significa que la correspondencia entre el scientificName del conjunto de datos y el del árbol taxonómico es completa.
FUZZY: implica que la correspondencia entre el scientificName del conjunto de datos y el del árbol taxonómico es parcial. Probablemente, el nombre difiere en su escritura, lo que indica errores de tipeo o diferencias por correcciones nomenclaturales. Por ejemplo, la terminación “i” o “ii” cuando la especie se dedica a una persona.
HIGHERRANK: significa que la correspondencia entre el nombre científico del conjunto de datos y el del árbol taxonómico fue parcial. No se identificó el scientificName del taxón, pero sí en un nivel superior. Por ejemplo, si el scientificName corresponde a una especie, la correspondencia con el árbol taxonómico de GBIF fue a nivel de género. Esto sucede cuando el taxón aún no está en el árbol taxonómico de GBIF o cuando hay errores de tipeo mayores.
NONE y BLANK: implica que la correspondencia entre el scientificName del conjunto de datos y el del árbol taxonómico fue nula o hubo varias coincidencias con muy poca información para determinar un resultado. Esto sucede cuando hay homónimos o cuando el taxón aún no se encuentra en el árbol taxonómico de GBIF, como es el caso de especies recientemente descritas o algunas endémicas.
Si los siguientes elementos están documentados en el conjunto de datos original, tendrán la tripleta mencionada: scientificNameAuthorship, kingdom, phylum, class, order, family, genus y specificEpithet.
4.2 Datos de grupos biológicos Marino Costeros
Funcionamiento:
Esta rutina está diseñada para ser implementada en conjuntos de datos de grupos biológicos marinos, ya que emplea como fuente de referencia los taxones marinos del árbol taxonómico de LifeWatch (LW-SIBb) a través de un llamado al API de WoRMS (World Register of Marine Species). La rutina retorna la taxonomía superior, nombres aceptados, estatus taxonómico y autoría del nombre científico de acuerdo al árbol taxonómico mencionado y los compara con los elementos documentados en el archivo base, generando los indicadores de validación.
En adición a los elementos taxonómicos, esta rutina retorna otros elementos útiles que brindan información sobre el tipo de hábitat del taxón y el LSID de WoRMS o AphiaID, elemento requerido para la publicación de datos a través de OBIS (Ocean Biodiversity Information System).
Resultados:
En las primeras columnas del proyecto, encontrará una columna con el valor taxonómico original, un valor por el al árbol taxonómico y el indicador de validación que establece si los valores coinciden (1) o difieren (0), como se muestra en la las rutinas previas (Fig. 7).
A continuación, se listan las columnas que encontrará después de ejecutar la rutina, sin incluir las que ya se mencionaron en las rutinas previas de validación taxonómica (Fig. 8):
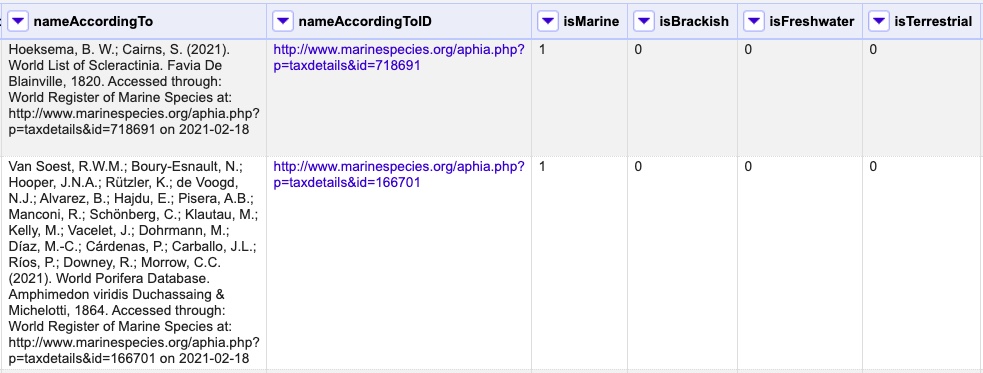
Figura 8. Algunas columnas adicionales resultantes del proceso de validación con la rutina de WoRMS.
“matchType”: es el resultado del cruce de los datos originales con el árbol taxonómico de WoRMS a partir del elemento scientificName. Los valores que encontrará en esta columna son:
exact: significa que la correspondencia entre el scientificName del conjunto de datos y el del árbol taxonómico es completa.
phonetic: implica que la correspondencia entre el scientificName del conjunto de datos y el del árbol taxonómico es completa a nivel fonético, a pesar de algunas diferencias menores en la escritura.
near_1: hay una diferencia de un carácter entre el scientificName del conjunto de datos y el del árbol taxonómico. Es una correspondencia bastante confiable.
near_2: hay una diferencia de dos caracteres entre el scientificName del conjunto de datos y el del árbol taxonómico. Se sugiere una revisión del nombre.
near_3: hay una diferencia de tres caracteres entre el scientificName del conjunto de datos y el del árbol taxonómico. Se requiere una revisión del nombre.
Otras posibilidades poco frecuentes: match_quarantine y match_deleted. En estos casos, WoRMS recomienda contactarlos directamente.
5.1 Datos de grupos biológicos principalmente continentales
Realice un Facet en “taxonMatchType” e identifique los posibles errores señalados como FUZZY y NONE. Generalmente, este último se presentará si no realizó una limpieza de los calificadores de identificación como “cf.” y “sp.” en el scientificName antes de ejecutar la rutina. Por lo tanto, es pertinente que realice los ajustes en esos nombres científicos según corresponda.
Realice la limpieza de los FUZZY, teniendo en cuenta que algunos de los nombres son sinónimos (lo cual puede verificar en el elemento “taxonomicStatusSuggested”. Deje el nombre aceptado para este caso y haga los respectivos cambios en scientificName, al igual que en genus y specificEpithet, segun corresponda, para que tengan coherencia con el scientificName.
Realice un Facet en los elementos de validación como “classValidation” y “familyValidation”. Seleccione el buleano 0 e identifique y ajuste los errores que le muestre la validación.
Complete la información para la taxonomía superior incompleta (phylum, order, genus y specificEpithet) de acuerdo a los campos sugeridos (“suggested”) por la tripleta de resultados.
Complete el elemento taxonRank a partir del “suggestedTaxonRank”, teniendo en cuenta el vocabulario controlado para este elemento.
En OpenRefine puede ir a la columna que quiere completar (por ejemplo, phylum), seguir la ruta Edit cells>Transform y escribir la siguiente expresión en el recuadro de edición: cells[‘phylumSuggested’].value. De esta manera, el programa remplazará el contenido vacío por el sugerido en “phylumSuggested”. Haga lo mismo para los demás elementos, cambiando la expresión según corresponda.
5.2 Datos de grupos biológicos marino-costeros
Revise la columna “matchType” e identifique los nombres científicos que no coinciden con el árbol taxonómico de WoRMS. Posteriormente, ajústelos según corresponda.
Revise las columnas adicionales que trae el script y examine las tripletas de validación haciendo Facet en estos elementos con OpenRefine y seleccione la opción 0. Por último, identifique dónde hay inconsistencias y corríjalas.
Reto: ¿Hay registros que sean completamente terrestres y no del medio marino según la validación?. Identifique el error en la validación con los elementos “isMarine”, “isBrackish”, “isFresh”, “isTerrestrial”.
Compare sus resultados con los siguientes archivos, validados según las definiciones del estándar Darwin Core, para las rutinas con el API de GBIF y WoRMS, identificando aciertos y oportunidades de mejora.
¿Qué diferencias encontró con sus resultados?
«Archivo validado para datos continentales».
«Archivo validado para datos marino-costeros».
Si tiene datos propios que desea publicar, intente realizar la validación correspondiente con las rutinas para datos continentales o marino-costeros.
¡Felicitaciones! Ha finalizado la validación taxonómica con GBIF (Datos continentales) y WoRMS (Datos marino-costeros), utilizando las rutinas de validación en OpenRefine que fueron desarrolladas por el EC-SiB Colombia.
Desarrollada para estandarizar los contenidos de los elementos de la geografía superior, especialmente stateProvince, county y municipality con base en una fuente de referencia nacional. La rutina contrasta los valores documentados con la información oficial para Colombia, a partir de un archivo de referencia previamente cargado en OpenRefine, y genera indicadores de validación. Los indicadores permiten establecer dos tipos de errores en la geografía superior: errores de tipeo y gramática y errores de consistencia relacionados con la correspondencia entre entidades geográficas, como municipios (county) o centros poblados (municipality), que no pertenecen al departamento (stateProvince).
El archivo oficial de referencia del repositorio se genera con la información geográfica de la División Político Administrativa definida por el DANE (Divipola). Vale la pena precisar que esta rutina puede implementarse para otros países, pero con la información geográfica oficial del país de interés y empleando la misma estructura del archivo mencionado para Colombia.
Datos de validación
Cree un proyecto en OpenRefine a partir del conjunto de datos «datos_ValidacionGeografia.xlsx» y asígnele el nombre de datos_ValidacionGeografia. Si tiene dudas sobre cómo hacerlo, revise el paso 2 de la guía general de OpenRefine.
Asegúrese de que el conjunto de datos o los elementos a validar estén estructurados según el estándar Darwin Core (stateProvince, county y municipality).
Para ejecutar esta rutina, es necesario que cargue el archivo «DIVIPOLA_2021-0416.xls» en OpenRefine. Si tiene dudas sobre cómo hacerlo, revise el paso 2 de la guía general de OpenRefine.
Luego, nombre el proyecto en OpenRefine como DIVIPOLA_20210416 (Fig. 9).
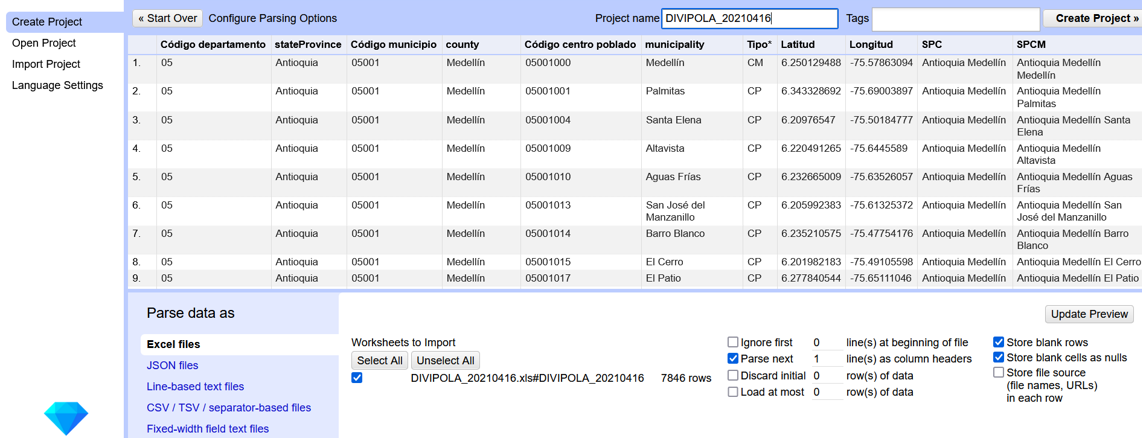
Figura 9. Carga de archivo DIVIPOLA 20210416 en OpenRefine.
4.1. Copiado de la rutina
Diríjase a la rutina de Validación de nombres geográficos con DIVIPOLA. Será redirigido a GitHub, donde encontrará un archivo de texto plano. Copie el texto de la rutina de validación (Fig. 10), asegurándose de seleccionar solo la rutina -sin las instrucciones- y de copiar todos los corchetes iniciales ({) y finales (}), ya que estos son fundamentales para que la rutina se ejecute correctamente.
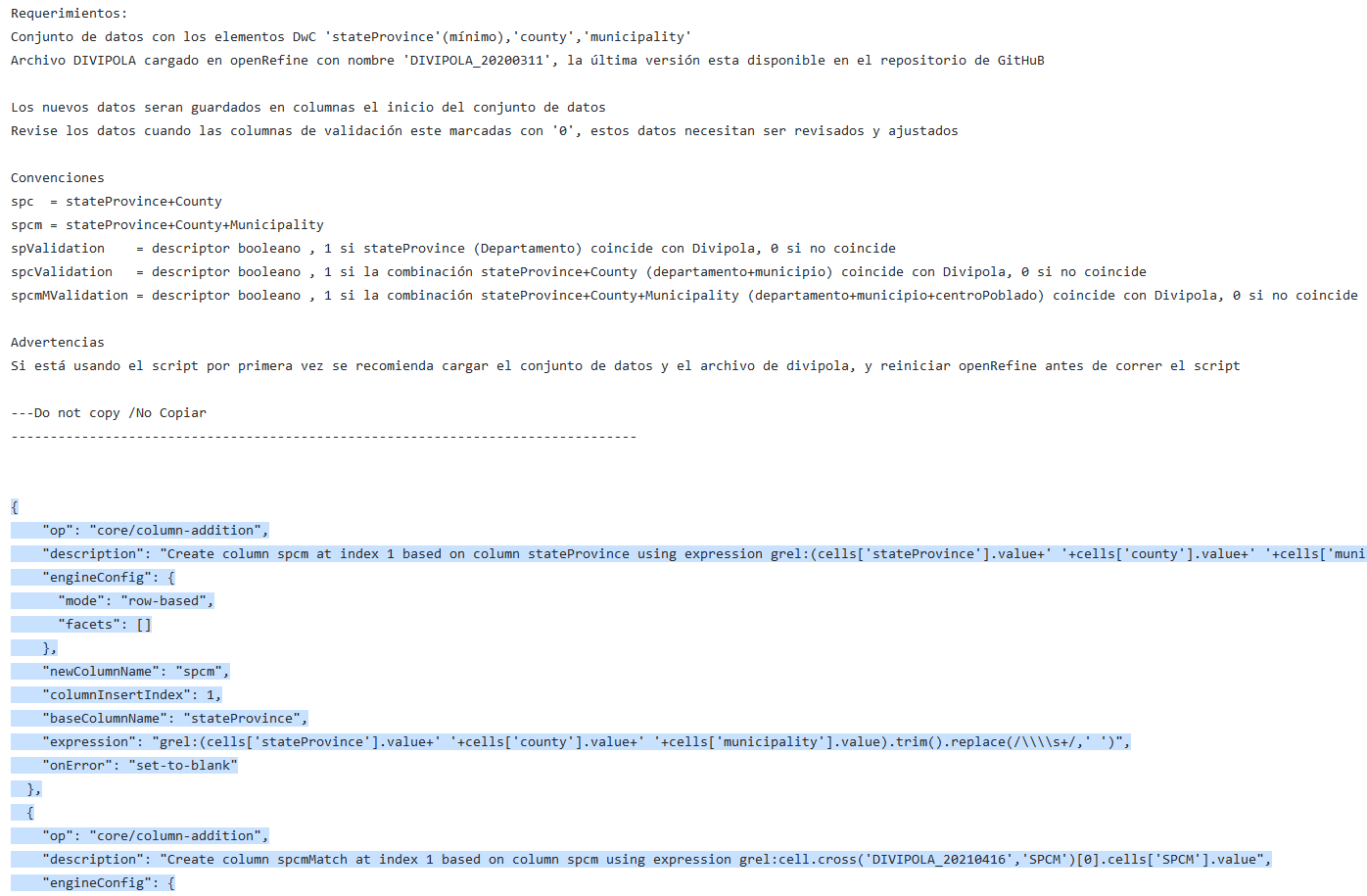
Figura 10. Copia de la rutina en GitHub.
4.2. Ejecución de la rutina
En el conjunto de datos a validar en OpenRefine («datos_ValidacionGeografia.xlsx»), diríjase al menú superior izquierdo, seleccione la pestaña Deshacer/Rehacer y haga clic en el botón Aplicar…. A continuación, se abrirá una ventana de texto vacía. Pegue la rutina a ejecutar en el cuadro de texto y dé clic en Ejecutar Operaciones (Fig. 11).
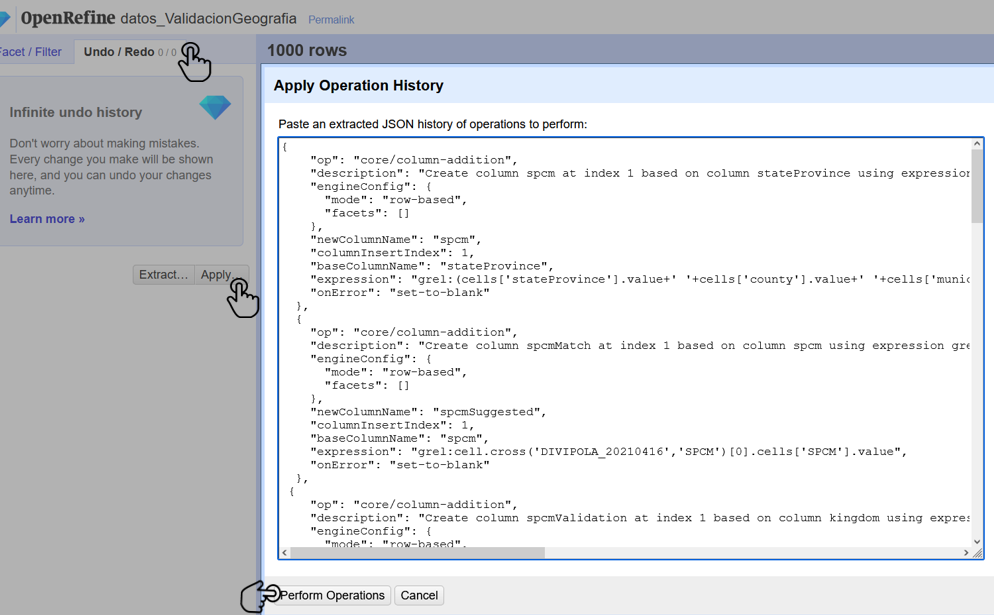
Figura 11. Pasos para la ejecución de la rutina en OpenRefine.
Espere a que finalice la ejecución de la rutina. Particularmente, esta rutina necesita consultar información en otro proyecto en OpenRefine, por lo que no depende de la conexión a Internet, pero su tiempo de ejecución varía según el número de filas del conjunto de datos y de la memoria RAM de su equipo.
Luego de ejecutar el script, algunas columnas adicionales se añadirán al conjunto de datos. El contenido de dichas columbas se describe a continuación:
“spValidation”: resultado de la validación entre el stateProvince y el de departamento del archivo Divipola. El resultado será 1 si hay coincidencia y 0 si no la hay.
“spc”: unión de los elementos stateProvince y county del conjunto de datos para realizar la validación por municipios.
“spcValidation”: resultado de la validación para stateProvince y county respecto al departamento y municipio del archivo Divipola. El resultado será 1 si hay coincidencia y 0 si no la hay.
“spcm”: unión de los elementosstateProvince, county y municipality del conjunto de datos para realizar la validación de centros poblados.
“spcmValidation”: resultado de la validación para stateProvince, county y municipality respecto al departamento, municipio y centro poblado del archivo Divipola. El resultado será 1 si hay coincidencia y 0 si no la hay.
Paso 5.1. Ajuste de los departamentos
Realice un Text facet en las columnas stateProvince y “spValidation”. En el Text facet de “spValidation”, seleccione los resultados no coindicentes 0 (Fig. 12).
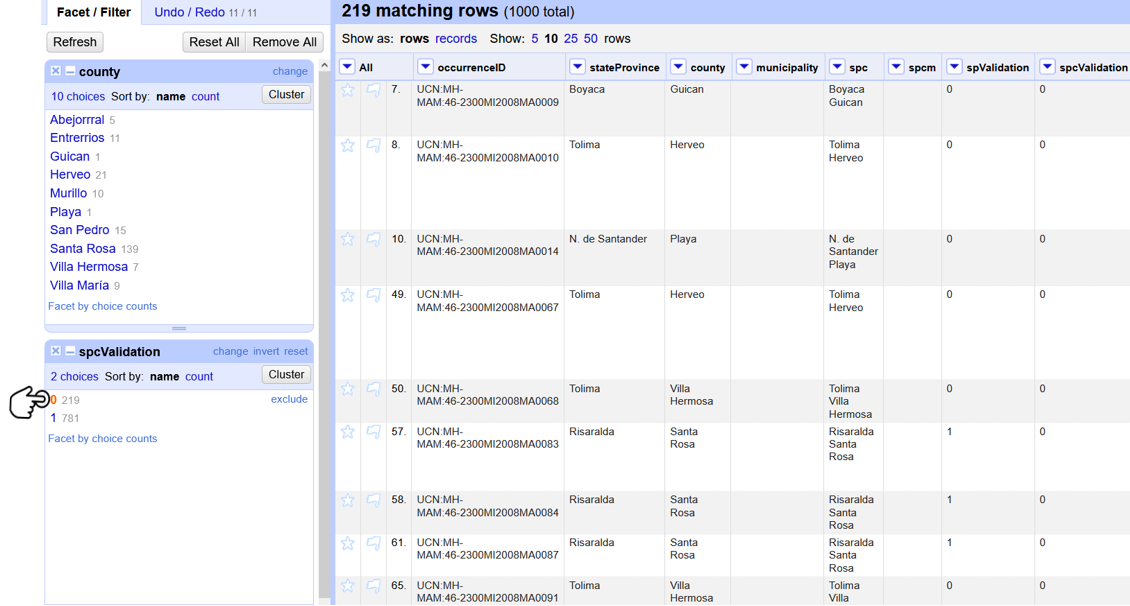
Figura 12. Text facet en OpenRefine para identificar los registros donde debe hacer ajustes sobre el elemento stateProvince.
Revise los departamentos en stateProvince y haga los ajustes que considere necesarios con base en el archivo de «DIVIPOLA_20210416».
Paso 5.2. Ajuste de los municipios
Ahora realice un Text facet en el elemento county y el elemento “spcValidation”. En el Facet, seleccione los resultados no coincidentes 0 (Fig. 13).

Figura 13. Text facet en OpenRefine para identificar los registros donde debe hacer ajustes sobre el elemento county.
Revise los municipios en county y haga los ajustes que considere necesarios según el archivo «DIVIPOLA_20210416».
Compare sus resultados con el siguiente archivo, validado según las definiciones del estándar, e identifique aciertos y oportunidades de mejora. ¿Qué diferencias encontró con sus resultados?
Realiza un llamado al API de GeoNames (servicio SRTM-1) a partir de los elementos Darwin Core de latitud (decimalLatitude) y longitud (decimalLongitude) en grados decimales. En este sentido, retorna la elevación con una resolución de 30 metros por pixel y la compara con los elementos documentados en el archivo base, generando los indicadores de validación.
Para esta parte, utilice la rutina Validación y recuperación de elevaciones a partir del API de Geonames
Descargue el archivo «datos_ValidacionElevaciones.xlsx» y cárguelo en OpenRefine. Si tiene dudas sobre cómo hacerlo, revise el paso 2 de la guía general de OpenRefine.
El conjunto de datos a validar debe tener como mínimo los elemento DwC decimalLatitude y decimalLongitude documentados adecuadamente. De lo contrario, la rutina no se ejecutará adecuadamente.
Si cuenta con elevaciones, es importante que estas estén documentadas en el elemento minimumElevationInMeters. Si están documentadas solo como verbatimElevation, el script solo traerá las elevaciones sugeridas y no habrá validaciones con las elevaciones documentadas.
Para este paso, es necesario tener una cuenta activa en GeoNames. Si no tiene una, regístrese aquí antes de correr la rutina.
Es muy importante tener en cuenta las siguientes indicaciones para habilitar su cuenta y poder usar los servicios web.
1) Cree su cuenta con un correo electrónico y contraseña. Asegúrese de que el correo esté bien escrito y recuerde su nombre de usuario, ya que será necesario para los siguientes pasos (Fig. 14).
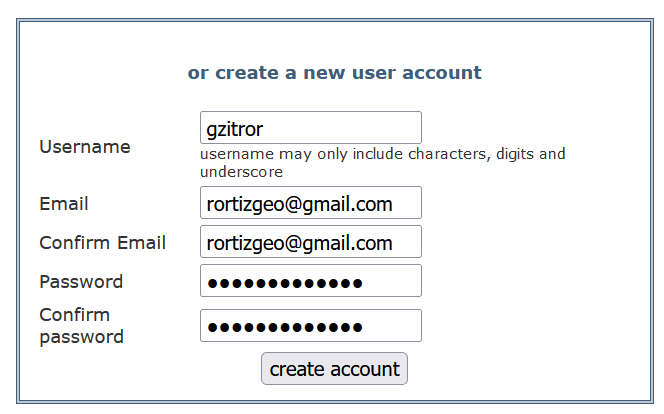
Figura 14. Creación de usuario en Geonames.
2) Abra el mensaje que recibirá en el correo que usó para el paso anterior (es posible que llegue a la carpeta de Spam) y haga clic en el link de confirmación (Fig. 15).
Figura 15. Correo de confirmación de Geonames.
3) En la esquina superior derecha, dé clic en su nombre de usuario. En la parte inferior, verá un mensaje indicando que la cuenta aún no está activada para hacer uso de los servicios web gratuitos. Haga clic en Click here to enable para activarlos (Fig. 3).
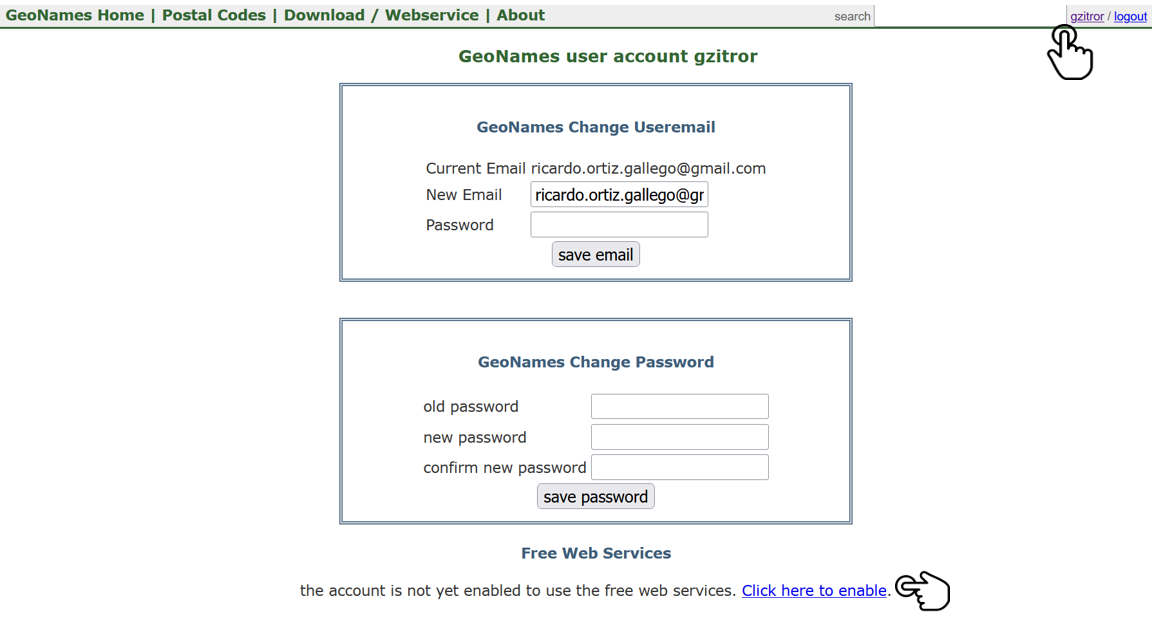
Figura 16. Habilitar opciones de uso de servicios web gratuitos de Geonames.
4) Recibirá un mensaje que confirma la habilitación de los servicios web para su cuenta (Fig. 17).

Figura 17. Mensaje de confirmación de habilitación de servicios web.
4.1 Copiado de la rutina
Diríjase a la rutina de Validación y recuperación de elevaciones a partir del API de Geonames. Será redirigido a GitHub, donde encontrará un archivo de texto plano. Copie el texto de la rutina de validación (Fig. 18), asegurándose de seleccionar solo la rutina -sin las instrucciones- y de copiar todos los corchetes iniciales ({) y finales (}), ya que estos son fundamentales para que la rutina se ejecute correctamente.
Figura 18. Copia de la rutina en repositorio en GitHub.
4.2 Ajuste de la rutina
Antes de ejecutar la rutina, remplace la palabra demo en la expresión username=demo por el nombre de su usuario en GeoNames. Por ejemplo, username=gzitror. Para ello, abra un editor de texto como el Bloc de notas de Windows y dé clic la opción Remplazar… del menú Edición. Luego, introduzca demo en el cuadro de texto “Buscar:” y escriba su usuario en el cuadro de texto “Reemplazar por:” (Fig. 19).
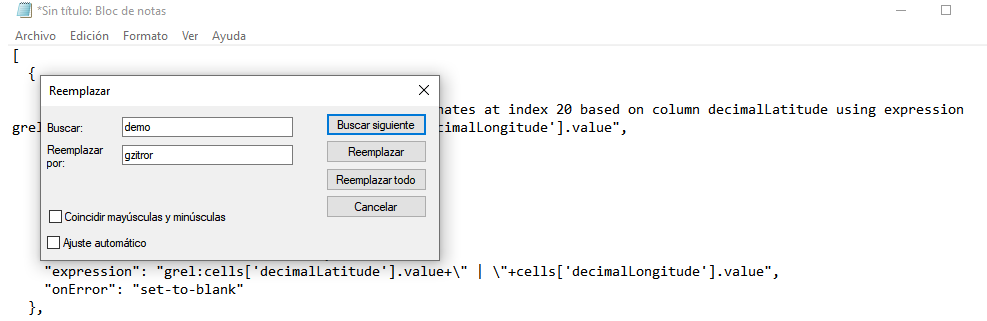
Figura 19. Busqueda y reemplazo del nombre de usuario para ejecutar la rutina usando el Bloc de notas de Windows.
Si ejecuta la rutina sin hacer este cambio, incorporará por defecto la opción de prueba (demo). Esta opción tiene un límite de 20.000 consultas diarias mundiales, por lo que es posible que el servicio esté agotado y no obtenga resultados.
Esta rutina captura la elevación a partir de las coordenadas decimales documentadas en los elementos DwC decimalLatitude y decimalLongitude del archivo base, a través de una consulta a los servicios de GeoNames. Además, se ejecuta sobre valores únicos de pares de coordenadas para evitar superar el límite de consultas diarias por usuario.
La rutina de elevaciones utiliza el modelo de elevación SRTM-1 (“srtm1”) por defecto, el cual cuenta con una resolución aproximada de 30 metros. Sin embargo, el usuario puede usar otro de los modelos de elevación disponibles:
SRTM3 (srtm3): datos de elevación de la Shuttle Radar Topography Mission (SRTM). Tienen una resolución aproximada de 90 x 90 metros.
Astergdemv2 (astergdem): datos de elevación del Aster Global Digital Elevation Model V2 (2011). Tienen una resolución aproximada de 30 x 30 metros.
GTOPO30 (gtopo30): modelo de elevación global con resolución aproximada de 30 arcos por segundo. Es equivalente a una grilla de 1 km x 1 km.
Para cambiar el modelo de elevación, reemplace el valor srtm3 en la expresión grel "http://api.geonames.org/srtm1 por el valor que corresponda al servicio que desea utilizar (astergdem o gtopo30 en la rutina. En este ejercicio, se utiliza el modelo por defecto: srtm3.
4.3. Ejecución de la rutina
En el conjunto de datos a validar en OpenRefine («datos_ValidacionElevaciones.xlsx»), diríjase al menú superior izquierdo, seleccione la pestaña Deshacer/Rehacer y haga clic en el botón Aplicar…. A continuación, se abrirá una ventana de texto vacía. Pegue la rutina a ejecutar en el cuadro de texto y dé clic en Ejecutar Operaciones (Fig. 20).
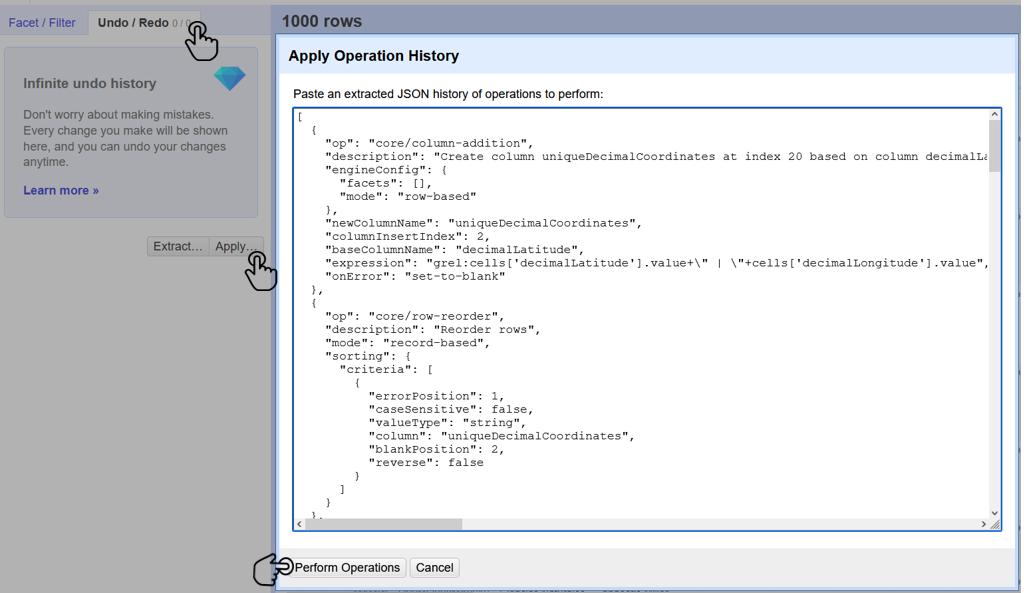
Figura 20. Pasos para la ejecución de la rutina en OpenRefine.
5.1 Interpretación de resultados
En las primeras columnas del proyecto, encontrará los elementos de elevación reorganizados y las tres columnas de validación obtenidas de la rutina (Fig. 21).
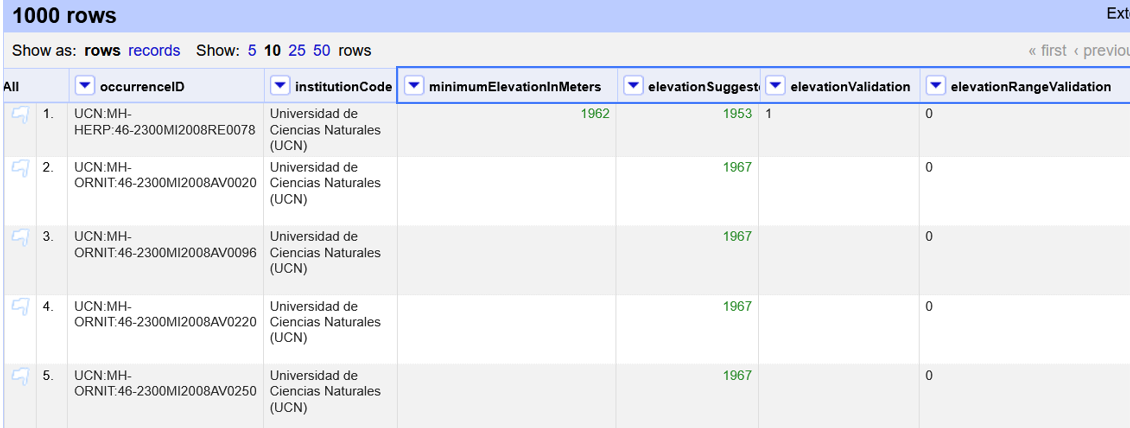
Figura 21. Columnas resultantes de la validación en OpenRefine.
Encontrará, de manera intercalada, las columnas originales, un valor sugerido por el servicio de elevación (“elevationSuggested”) y dos indicadores de validación (Figura 21):
El indicador “elevationValidation“ compara el valor registrado en el elemento minimumElevationInMeters con lo sugerido por servicio de elevación. El resultado debe ser interpretado así:
1: indica que la diferencia entre la elevación documentada en el minimumElevationInMeters y el valor sugerido en “elevationSuggested” es menor a 100 m.
0: significa que diferencia entre la elevación documentada en el minimumElevationInMeters y el valor sugerido en “elevationSuggested” es mayor a 100 m.
No hay elevación mínima documentada.
El indicador _“elevationRangeValidation” contrasta compara el rango registrado en los elementos minimumElevationInMeters y maximumElevationInMeters con lo sugerido por el servicio de elevación. El resultado debe ser interpretado así:
1: indica que el rango de elevaciones contiene la elevación sugerida.
0: significa que el rango de elevaciones no contiene la elevación sugerida.
La validación de rangos de elevaciones en ocasiones suele presentar inconvenientes. El equipo del SiB Colombia está ajustando el script para facilitar su uso.
5.2 Revisión de los resultados
Compare sus resultados con el siguiente archivo, validado según las definiciones del estándar, e identifique aciertos y oportunidades de mejora. ¿Qué diferencias encontró con sus resultados?
Si tiene datos propios que desea publicar, intente realizar la validación correspondiente con las rutinas y procure incorporarlas a su flujo de trabajo.
¡Felicitaciones! Terminó el laboratorio de uso avanzado de OpenRefine.
Atribución y uso de los laboratorios

La licencia CC-BY permite usar, redistribuir y construir sobre estos contenidos libremente.
¡La difusión de estos laboratorios contribuirá a la publicación de más y mejores conjuntos de datos sobre biodiversidad!
Citación sugerida
Plata C., Ortíz R., Marentes E., Lozano J.(2021). Laboratorio de datos, Ciclo de formación. Consultado a través del SiB Colombia. Disponible en https://biodiversidad.co/formacion/laboratorios.
Este sitio web usa cookies, algunas son técnicamente necesarias otras mejoran la experiencia de usuario. Puede rechazar las cookies no esenciales seleccionando “Rechazar”. Consulte la Política de privacidad del sitio web para obtener más información.